Grüne Gentechnologie
DNA-Rekombination und Grüne Biotechnologie
Die DNA-Rekombination stellt die Grundlage für die stabile genetische Transformation dar, bei der neu in eine Zelle eingebrachte Sequenzen mit der Erbinformation verknüpft werden. Grundsätzlich unterscheidet man bei diesen Vorgängen zwischen homologer Rekombination und nicht-homologer Rekombination. Die homologe Rekombination ist insbesondere biotechnologisch von großem Interesse, worauf weiter unten noch eingegangen wird.
Molekulare Analyse von Rekombinations-Ereignissen – ortsspezifische DSBs
Zur detaillierten Analyse der Doppelstrangbruch-Reparaturmechanismen und der zugrunde liegenden Rekombinationsvorgänge ist es notwendig, in einem Genom an einer genau definierten Stelle einen Strangbruch zu erzeugen. Nur so kann man hinterher mit molekularbiologischen Techniken (PCR , Sequenzierung) die reparierte Stelle analysieren und sich ein Bild davon machen, wie die Enden wieder miteinander verknüpft worden sind.
Wie erzeugt man jedoch an einer einzigen Stelle im Genom einen solchen Bruch?
Die für Klonierungsarbeiten oft verwendeten Restriktions-Enzyme sind dafür nicht geeignet – sie würden in einem Genom wie dem von Arabidopsis viel zu viele Erkennungssequenzen schneiden. Was man also benötigt, ist im Prinzip eine hochspezifische molekulare Schere. In der Tat gibt es ein solches Werkzeug: die Megaendonuklease I-SceI , (Abbildung 1), ein Enzym was in der Natur in der Bäckerhefe (Saccharomyces cerevisiae) vorkommt.
Was prädestiniert dieses Enzym als molekulare Schere?
Die Erkennungssequenz von I-SceI hat eine Länge von 18 Nukleotiden – bei 4 verschiedenen Möglichkeiten pro Position ergeben sich weit mehr als 68 Milliarden Kombinationen – dass genau diese Sequenz also im Arabidopsis-Genom mit einer Größe von etwa 130 Millionen Basenpaaren vorkommt, ist also nicht sehr wahrscheinlich. Tatsächlich findet man in der Arabidopsis-Genom-Datenbank diese Sequenz nicht.
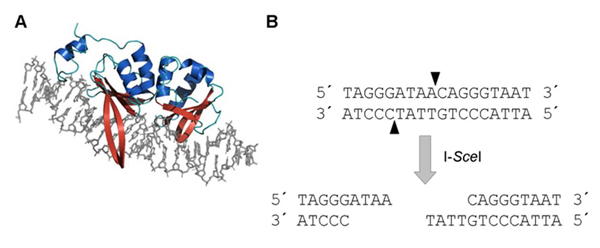
Abbildung 1: Die Meganuklease I-SceI. (A) Darstellung des an doppelsträngige DNA gebundenen I-SceI (mit freundlicher Genehmigung von CELLECTIS SA), (B) Erkennungssequenz von I-SceI. Die 18 Nukleotide umfassende Erkennungssequenz wird von I-SceI geschnitten, wobei 3´-Überhänge von 4 Nukleotiden Länge entstehen. Auf Grund der Länge der Erkennungssequenz ist es sehr unwahrscheinlich, dass diese zufällig in einem Genom wie dem von Arabidopsis auftritt.
Im Labor ist es möglich, sozusagen maßgeschneiderte T-DNAs herzustellen und mit Hilfe von Agrobakterien in Pflanzen zu transferieren. Auf diese Weise können transgene Pflanzen erhalten werden, die z.B. eine oder zwei Erkennungssequenzen für I-SceI haben. Sorgt man in diesen Pflanzen dafür, dass die Meganuklease vorhanden ist, können an genau bekannten Stellen Doppelstrangbrüche herbeigeführt werden. Daraus ergeben sich verschiedene Anwendungen – einige davon werden hier kurz dargestellt:
Entfernung von Markergenen
Die stabile genetische Transformation von Pflanzen mit Hilfe von Agrobakterien ist inzwischen eine Standardmethode – allerdings werden bei weitem nicht alle Zellen transformiert, so dass es notwendig ist, eine positive Selektion durchzuführen. Dies erfolgt in der Regel durch Selektionsmarkergene, die oft eine Resistenz gegen bestimmte Antibiotika vermitteln. Genau dies ist einer der Gründe, weshalb die Akzeptanz gentechnisch modifizierter Pflanzen in der Bevölkerung recht gering ist – es besteht die Sorge, dass beim Verzehr genetisch modifizierter Nahrungsmittel Teile dieser Transgen-Sequenzen u.a. mit dem Antibiotika-Resistenzgen von Bakterien der Darmflora aufgenommen werden könnten und dadurch Antibiotika-resistente Stämme entstehen könnten. Tatsächlich konnte ein solcher Vorgang bisher in keiner wissenschaftlichen Studie belegt werden, dennoch wäre es zur Erhöhung der Akzeptanz solcher Pflanzen bzw. der von ihnen produzierten Nahrungsmittel wünschenswert, Markergene wieder zu entfernen, sobald sie ihre Aufgabe erfüllt haben. Dass dies in der Tat möglich ist, konnte erstmals durch in unserer Arbeitsgruppe gezeigt werden (Abbildung 2, Siebert & Puchta, 2002).
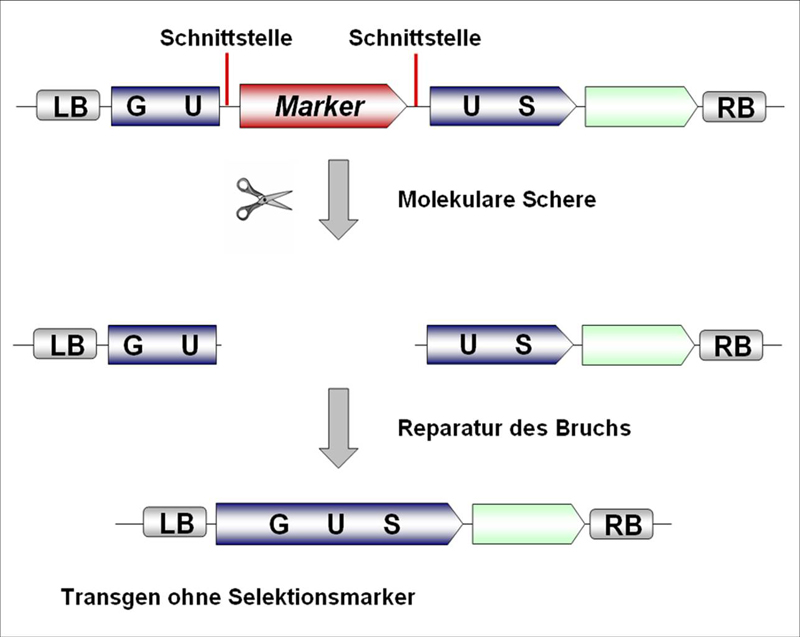
Abbildung 2: Eliminierung von Markergenen. Durch die Verwendung der Meganuklease I-SceI als Molekulare Schere konnte erstmals gezeigt werden, dass ein Markergen in lebenden Zellen ausgeschnitten werden kann und der daraus resultierende Doppelstrangbruch in der DNA wieder repariert wird – in diesem Falle durch homologe Rekombination zwischen den identischen Teilsequenzen des GUS-Reportergens (U). Diese Reparaturereignisse lassen sich histochemisch nachweisen.
Gezielter Austausch von Kopplungsgruppen
Bei der klassischen Pflanzenzüchtung werden zufällige Rekombinations-Vorgänge zwischen wünschenswerten Merkmalen der Elterngenome identifiziert und selektiert. Es ist jedoch sehr zeitaufwändig, auf diese Weise Pflanzen mit den gewünschten Eigenschaften zu erhalten. Ein Ansatz zur Beschleunigung dieses Prozesses ist die beabsichtigte Herbeiführung von zwei simultanen Doppelstrangbrüchen in einem Genom: hierbei werden – vereinfacht ausgedrückt – von zwei Chromosomen Teilstücke abgeschnitten (Abbildung 3). Die Enden dieser Stücke werden als Doppelstrangbrüche erkannt und repariert – geschieht dies auf die „falsche“ Weise, so ist eine reziproke Translokation die Folge. Auf diese Weise können bestimmte Genkombinationen gezielt ge- oder entkoppelt werden, womit sich die Ziele der Züchtung – die Kombination wünschenswerter Merkmale – schneller erreichen ließen. Dass dieses Vorgehensweise in Pflanzen grundsätzlich möglich ist, konnte erstmals 2007 in unserer Arbeitsgruppe bei Experimenten mit Tabak gezeigt werden (Pacher et al., 2007).
.jpg)
Abbildung 3: I-SceI-vermittelte Induktion einer reziproken Translokation im Genom von Nicotiana tabacum. Auf verschiedenen Chromosomen (weiß, grau) im Tabak-Genom wurden zwei verschiedene Transgensequenzen (T-DNAs A—B und C—D) integriert. Beide T-DNAs enthielten eine Schnittstelle für die Meganuklease I-SceI (roter Balken). Durch Expression von I-SceI wurden gleichzeitig zwei Doppelstrangbrüche in einem Genom herbeigeführt. Es konnte gezeigt werden, dass die Reparatur dieser Brüche tatsächlich durch reziproke Translokationen stattfinden kann und damit zwei neue Kopplungen (A—D und C—B) auftreten.
Gene Targeting
Für die Beantwortung vieler Fragestellungen in der Grundlagenforschung sind Mutanten die Lösung – so kann man an ihnen direkt untersuchen, ob ein fehlendes Gen bzw. Protein zu einem gegenüber dem Wildtyp veränderten Phänotyp führt. Im Falle unseres Modell-Organismus sind bislang jedoch vorwiegend Insertions-Mutanten die Untersuchungsobjekte, bei denen die T-DNA aus Agrobakterien zufällig in ein Gen inseriert wurde. Oft werden Teile des Gens vor oder hinter der Insertionsstelle noch immer exprimiert und die dabei möglicherweise entstehenden Teilproteine könnten mit ihren Funktionen Beobachtungen beeinflussen und damit Ergebnisse verfälschen. Auch bei der Herstellung transgener Pflanzen-Linien kann eine T-DNA „aus Versehen“ ein Gen zerstören. Folglich wäre es wünschenswert, die Integration von DNA-Stücken in das Pflanzengenom besser kontrollieren zu können bzw. den Integrationsort exakt zu definieren (Gene Targeting). Das ist möglich – in dem das Transgen mit so genannten Homologien, d.h. identischen Sequenzbereichen zum Integrationsort ausgestattet wird – die Integration erfolgt über homologe Rekombination. Diese ist in den meisten Pflanzenzellen jedoch sehr ineffizient.
Wie kann man dies ändern? Eine Möglichkeit besteht darin, an der zu verändernden Stelle im Genom einen Doppelstrangbruch zu erzeugen, denn dieser muss repariert werden. Stellt man gleichzeitig ein DNA-Stück zur Verfügung, welches zu beiden Seiten des erzeugten Bruches Sequenzhomologien aufweist, lässt sich eine gezielte Integration begünstigen. Für Pflanzen konnte dies erstmals 1998 gezeigt werden (Salomon & Puchta, 1998). Einen Nachteil hat diese Methode jedoch noch: die Meganuklease I-SceI schneidet nur die vorgegebene Erkennungssequenz (Abbildung 1) – es können also keine Doppelstrangbrüche an jeder gewünschten Position im Genom erzeugt werden. Um diese Limitierung zu überwinden, bedarf es neuer molekularer Scheren – hierzu wird derzeit mit verschiedenen Kooperationspartnern begonnen, maßgeschneiderte Meganukleasen und auch sequenzspezifische Zinkfingernukleasen zu verwenden.
Eine weitere Variante zur Verbesserung der Gene Targeting-Technik in Pflanzen, an der wir zur Zeit arbeiten, ist folgende: für alle Rekombinations-Vorgänge sind zahlreiche Proteine erforderlich – manche wohl ausschließlich für die nicht-homologe Rekombination, andere wiederum nur für die homologe Rekombination und wieder andere spielen in beiden Prozessen eine Rolle. Durch unsere Arbeiten am Lehrstuhl werden u.a. Proteine aus Arabidopsis analysiert, die einen Einfluss auf die homologe Rekombination haben (RecQ-Helikasen, Brca-Gene). Sorgt man nun durch Transgene für eine Überexpression von Proteinen, die an der homologen Rekombination beteiligt sind, so könnte dies die orts-spezifische Integration von Transgenen erleichtern. Dies sind Arbeiten, die wir in aktuellen Projekten verfolgen und bei denen wir die Antwort noch nicht kennen …
Steigerung der Meiotischen Rekombination, ein neuer Weg der Pflanzen-Züchtung
Wie oben bereits angedeutet wurde, ist eines unserer Ziele die Begünstigung der ohnehin in der Meiose ablaufenden Neukombination der beiden elterlichen Merkmale. Wie in dem Artikel über Meiose beschrieben, werden hierfür ganz gezielt DNA-Doppelstrangbrüche durch spezialisierte Enzyme genau zu diesem Zeitpunkt herbeigeführt. Ein aktueller Ansatzpunkt unserer Forschung ist es, die Anzahl solcher Brüche in der Meiose zu erhöhen, in dem z.B. maßgeschneiderte Meganukleasen oder Zink-Finger-Nukleasen spezifisch während der Meiose exprimiert werden. Ein weiterer Ansatzpunkt besteht darin, Proteine in der Meiose überzuexprimieren, die für die Auflösung der Rekombinations-Strukturen zuständig sind. Zusammengefasst könnten beide Strategien den Austausch von Allelen während der Meiose verbessern helfen und damit die Gewinnung gewünschter Merkmalskombinationen bei der Kreuzung begünstigen. Um dieses Projekt zu verwirklichen, werden derzeit verschiedene Arbeitsgruppen und Firmen im Ausland sowohl unser Institut durch das 7. EU-Rahmenprogramm mit 3 Millionen Euro gefördert (Pressemeldung RECBREED).

