Proteinbiochemie
Warum untersuchen wir Proteine biochemisch?
Ein wichtiger Baustein, um die Funktion eines Proteins zu verstehen, ist es, dieses isoliert bzw. unter definierten Bedingungen zu untersuchen. In wie weit hilft es, die Funktion eines Proteins isoliert zu betrachten? Oft gibt es in Organismen mehrere Proteine mit sehr ähnlichen Funktionen. Wenn z.B. das Protein X ausfällt, könnten dessen Funktionen von anderen Proteinen übernommen werden, oder es werden alternative Wege eingeschlagen, die das X-Protein nicht benötigen, und es ist daher bei der Analyse des Gesamtorganismus schwer zu sagen, was das Protein X alles kann. Gerade, wenn man sehr ähnliche Funktionen erwartet, können die eventuell vorhandenen Unterschiede biochemisch herausgearbeitet und die Proteine so von einander abgegrenzt werden. Das bringt uns zweierlei. Zum Einen bekommen wir Hinweise auf mögliche funktionelle Spezialisierungen und zum Anderen können wir so Proteine aus verschiedenen Organismen vergleichen. Das wiederum erlaubt uns Aussagen über die Evolution der Proteine zu machen. Wollen wir uns das mal an einem Beispiel anschauen.In unserer Modellpflanze Arabidopsis thaliana gibt es sieben verschiedene RecQ-Helikasen, im Menschen z.B. gibt es fünf verschiedene (Link). In der Bäckerhefe aber, gibt es zum Beispiel nur eine RecQ Helikase. Erst mal weiss man nicht, welche Aufgaben die pflanzliche RecQ Helikase Y in der Zelle übernimmt. Außerdem ist es ja denkbar, dass die menschliche RecQ Helikase Z die gleichen Funktionen hat, wie das pflanzliche Y. Wie ist das zum Beispiel mit AtRECQ2 und AtRECQ3?
Wie untersuchen wir Proteine biochemisch?
Da die Proteine der DNA-Rekombination und –Reparatur, die uns interessieren, meist nur in geringen Konzentrationen in der Pflanzenzelle vorkommen, bringen wir den Bauplan für das pflanzliche Protein in z.B. das Bakterium E. coli ein, das dann eine große Menge des zu untersuchenden Proteins produziert, wir nennen das dann „Expression“. Zusätzlich erweitern wir die Proteine oft um einen sogenannten „Tag“, der es uns später erleichtert, spezifisch unser gewünschtes Protein von den bakteriellen Proteinen abzutrennen, es also zu reinigen.
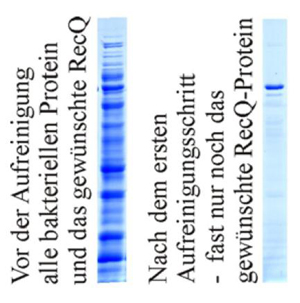
Abbildung 1: Coomassie-Färbung zweier Spuren eines SDS-PAGE Gels, das sämtliche Proteine (von oben nach unten) gemäß ihrer Größe auftrennt. Zu sehen ist der Erfolg der ersten Aufreinigung von AtRECQ3 mit einem His-Tag über eine sogenannte Ni-IMAC.
Nachdem wir mit Hilfe von Kontrollen sichergestellt haben, dass wir nur die Aktion des gewünschten Proteins betrachten, können wir jetzt Strukturen, wie sie in der DNA Reparatur und –Rekombination vorkommen, herstellen, und schauen, was unsere Proteine damit anstellen. So können wir z.B. sogenannte Holliday Junctions herstellen, die im Prozess der Homologen Rekombination eine wichtige Rolle spielen.
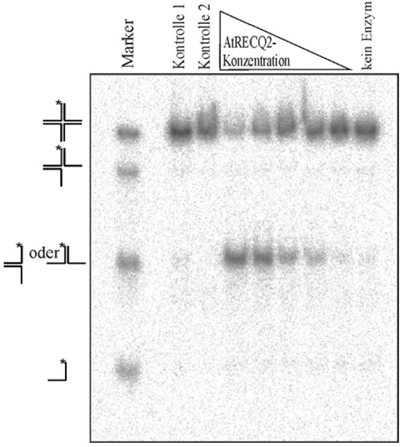
Abbildung 2: Analyse des Einflusses von AtRECQ2 auf eine Holliday Junction (schematisch oben links abgebildet). Dargestellt ist ein Autoradiogramm. Die verschiedenen DNA-Strukturen wurden durch eine native Gelelektrophorese voneinander getrennt (siehe Marker). Dort wo der Stern eingemalt ist, ist eine radioaktive Markierung, und alles, was diese Markierung beinhaltet, kann man visualisieren. Es ist deutlich zu erkennen, dass AtRECQ2 die Holliday Junction prozessieren kann. Und zwar entstehen dabei zwei Gabelungen. Diese deuten auch auf eine so genannte Branch-Migration-Aktivität von AtRECQ2 hin.
AtRECQ2 (siehe Abbildung 2) - aber nicht AtRECQ3 - kann die Holliday Junction prozessieren, d.h. die beiden Proteine haben wahrscheinlich unterschiedliche Funktionen in der Zelle. Durch in vivo Analysen kann man nun genauer untersuchen, ob AtRECQ2 tatsächlich eine Rolle in der Homologen Rekombination spielt, wie es die biochemischen Analysen vorschlagen – ebenso werden jetzt die in vivo Daten von den biochemischen Daten gestützt. Durch eine Vielzahl von Experimenten haben wir herausgefunden, dass AtRECQ2 ähnliche biochemische Eigenschaften hat, wie die menschliche RecQ Helikase WRN. Wir können also im Hinblick auf die Evolution die Hypothese aufstellen, dass die beiden funktionelle Homologe sind. Interessante neue Erkenntisse über AtRECQ2 können dann evt. auch auf das menschliche WRN zutreffen. AtRECQ3 hingegen ist unter Umständen ein Protein, das kein menschliches Equivalent hat – hier haben wir also die Möglichkeit Aussagen über ein pflanzenspezifisches RecQ-Protein zu treffen.
Was machen wir konkret?
Wir analysieren nicht nur AtRECQ2 und AtRECQ3, sondern auch jede Menge anderer Helikasen, sowie DNA-Nukleasen oder DNA-Einzelstrangbindeproteine. Neben den reinen Charakterisierungen der Einzelproteine, schauen wir auch, wie sie sich gegenseitig beeinflussen. Weiterhin wollen wir auch über biochemische Methoden Interaktionspartner unserer Proteine identifizieren oder aber unser aufgereinigtes Protein nutzen, um spezifische Antikörper herstellen zu lassen, was uns wiederum den Zugang zu neuen interessanten Analysemethoden eröffnet.