| Titel | Bearbeiter | Typ | Datum |
|---|---|---|---|
| Morphological verification of genetic resources of Goji - Lycium barbarum and Lycium chinense and congeneric species - and development of DNA-based differentiation methods | Sascha Wetters | Masterarbeit | 2015 |
| Der wahre Goji? | Leonie Hacker, Laura Feller | F2 PE | 12.06.2015 |
| Der wahre Goji? | Stephanie Calin, Daniel Kuhn | F2 PE | 20.04.2015 |
Projekt Beschreibung

Als F3:
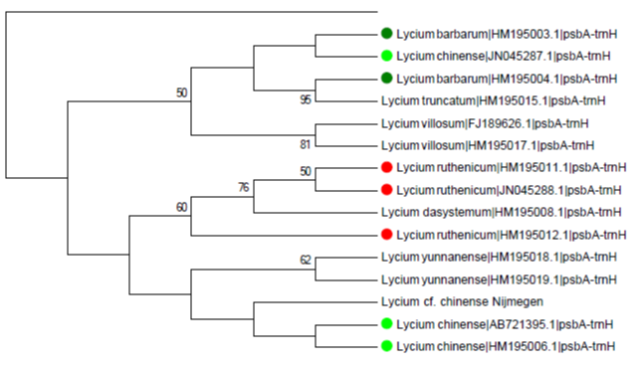
DNA Isolation aus getrocknetem Pflanzenmaterial von den verschiedenen Lycium Akzessionen. Qualitätskontrolle mittels Nanodrop und Gelelektrophorese. PCR Amplifikation von drei Barcoding Markern (matK, ITS, psbA-trnH). Kontrolle der Amplifikation und anschließenede Aufreinigung der Amplifikate für die Sequenzierung. Auswertung der Ergebnisse mit Hilfe von FinchTV. Erstellung von Phylogenetischen Bäumen mit MEGA und Untersuchung der Unterschiede auf Eignung für die Erstellung eines Authentifizierungsassays. Primer Design und Evaluation des Assays.
Molekulare Alternativen: DNA-Fingerprinting (RAPD, TBP, ISSR, APPCR)
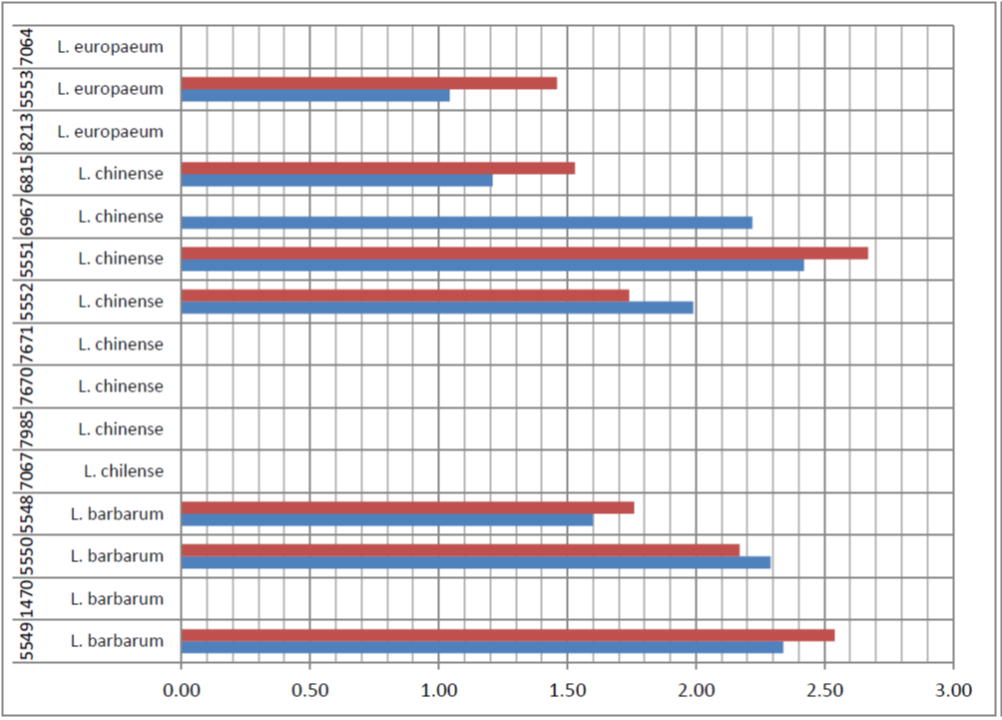
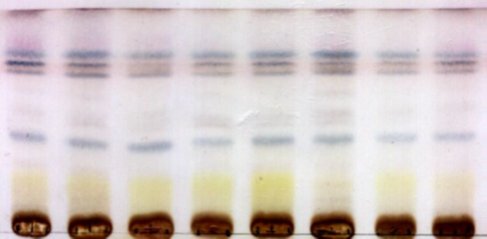
Alternativ oder bei Präferenz: Herstellung von Vollextrakten aus Früchten. Analyse der Extrakte mittels Dünnschichtchromatographie: HPTLC mit verschiedenen mobilen Phasen; Detektion mittels UV und anschließender Derivatisierung mit Anisaldehyd. Dokumentation der Ergenisse.
Als Bachelor Arbeit (ab August 2015):
Wie oben mit der Zusätzlichen Aufgabe der Bilddokumentation der im Garten blühenden und fruchtenden Akzessionen unter Einbeziehung eines Bestimmungsschlüssels. Anschließende Auswertung soll weitere Hinweise zur Identität der vorhandenen Akzessionen liefern. Bei Interesse kann versucht werden die Akzessionen auf deren Chromosomenzahl hin zu untersuchen. Mikroskopie der Blüten sowie Pollen steht ebenfalls zur Auswahl.
Hintergund Lektüre
T. Fukuda (2001). ‘Phylogeny and Biogeography of the Genus Lycium (Solanaceae): Inferences from Chloroplast DNA Sequences’. Molecular Phylogenetics and Evolution 19(2):246–258.
R. A. Levin & J. S. Miller (2005). ‘Relationships within tribe Lycieae (Solanaceae): paraphyly of Lycium and multiple origins of gender dimorphism’. American Journal of Botany 92(12):2044–2053.
R. G. Olmstead, et al. (1999). ‘Phylogeny and provisional classification of the Solanaceae based on chloroplast DNA’. Solanaceae IV 1(1):1–137.
O. Potterat (2010). ‘Goji (Lycium barbarum and L. chinense): Phytochemistry, pharmacology and safety in the perspective of traditional uses and recent popularity’. Planta Med 76:7–19.
T. Xin, et al. (2013). ‘Super food Lycium barbarum (Solanaceae) traceability via an internal transcribed spacer 2 barcode’. Food Research International 54(2):1699–1704.
X. Yao, et al. (2011). ‘Phytochemical and Biological Studies of Lycium Medicinal Plants’. Chemistry & Biodiversity 8(6):976–1010.
X.-l. Yin, et al. (2005). ‘Assessing phylogenetic relationships of Lycium samples using RAPD and entropy theory’. Acta Pharmacologica Sinica 26(10):1217–1224.
K. Y. B. Zhang, et al. (2001). ‘Differentiation of Lycium barbarum from its related Lycium species using random amplified polymorphic DNA’. Planta medica 67(4):379–381.